その予測モデル、ちゃんと予測できてますか?〜applicability domain の考慮〜
機械学習モデルを研究や開発の現場で活用しようとする試みが盛んですが、様々な要因で難航しているのではないでしょうか。
その一つとして、折角苦労して作ったモデルが役に立たない、ということが挙げられます。機械学習モデルを作ることが目的ではなく、(論文になる、特許を取れる、臨床試験を突破できる、商品として売れるなどで)役に立つ化合物を作ることが目的であるはずです。機械学習モデルもあくまで道具です。金槌は釘を打つのには役に立っても、目玉焼きを作るのには役に立たないように、使い方を考える必要があります。
機械学習モデルの使い方を考察するのに有用な概念として、適用範囲(applicability domain: AD)が挙げられます。
AD はその予測モデルがうまく機能する範囲、というのが大まかな考え方です。例えば、ベンゼンやトルエン、アニリン、フェノール、ブロモベンゼンなど、一置換型ベンゼンのデータを集めて物性データ予測モデルを作り、予測に使うことを考えます。クロロベンゼンの物性を予測したい場合、元のデータ群と構造が似ているので、直感的に上手く予想できそうです。しかし、エストラジオールなどのステロイド化合物をこのモデルで予測しようとしても、構造が違い過ぎるので、上手く予測できなさそうです。
より細かくは、データ密度の話になるのですが、その辺りの詳細な説明は、明治大学の金子先生の HP にあります。
実務上重要となるのは、AD 外にあるデータは予測出来ない可能性がある、ということです。予測データが AD 外にあるかを予め確認できれば、予測結果の解釈や次の実験を計画するのに役に立ちそうです。
そうなると、どのようにして AD を判別するかが重要になりそうです。
AD を判別する簡便な方法として、k 最近傍法、one class support vector machine (OCSVM)、アンサンブル学習が考えられます。これも詳細は金子先生の HP にあります。
今回は、OCSVM を使って AD を予測してみます。
import numpy as np import pandas as pd from sklearn.svm import OneClassSVM from sklearn.decomposition import PCA from sklearn.linear_model import LinearRegression from sklearn.metrics import r2_score, mean_squared_error import matplotlib.pyplot as plt import pickle # データの読み込み X_train = pd.read_csv("../data/LogS_train_scaled.csv", index_col = 0) X_test = pd.read_csv("../data/LogS_test_scaled.csv", index_col = 0) # 記述子名のリストを取り出す descriptor_label = X_train.columns y_train = pd.read_csv("../data/LogS_train_value.csv", index_col = 0) y_test = pd.read_csv("../data/LogS_test_value.csv", index_col = 0) # One class support vector machine (OCSVM) モデルを構築する clf = OneClassSVM(nu = 0.3) y_train_class = clf.fit_predict(X_train) y_test_class = clf.predict(X_test) # 分割面からの距離を算出 clf.decision_function(X_test)
OCSVM は scikit-learn に含まれているので、その実装は簡単です。
与えるパラメータとして、nu があります。これは大雑把に言うと、トレーニングデータの中でAD 外とみなすデータの割合です。
目的やデータにより、任意に決める必要がありますが、肌感覚的には、0.2~0.3 辺りを試すと良い気がします。AD を厳しく見積もりたい時は、もっと値を大きくしても良いと思います。
OCSVM はラベルが必要ない、教師なし学習ですので、fit_predict (fit) には説明変数だけを入力すれば良いです。
y_train_class と y_test_class は各データについて 1 か -1 の値が書かれたベクトルとして得られます。1 であれば AD 内、-1 であれば AD 外となります。
decision_function を使うと、AD の境界となる分割面からの距離を算出することができます。正負の関係は上と同様です。
データの分布と AD の関係を、主成分分析(PCA)の第一主成分と第二種成分を取り出してプロットしてみます。
# データの分布を主成分分析(PCA)で表現してみる pca = PCA() X_train_pca = pca.fit_transform(X_train) X_test_pca = pca.transform(X_test) # フォントを変更 plt.rcParams["font.family"] = "Arial" # Plot のインスタンスと図の大きさを定義 fig = plt.figure(figsize = (10, 10)) # インスタンスの中に複数のグラフを作る場合に指定 # 引数は(行数、列数、具体的に作りたい図のインデックス) # グラフを 1 つだけ作りたいときは (1, 1, 1)としてやれば良い ax = fig.add_subplot(1, 1, 1) # グラフのタイトル ax.set_title("Applicability domain", fontsize = 28) # グラフの表示範囲 ax.set_xlim(-15, 15) ax.set_ylim(-15, 15) # 目盛の数字の大きさを調整 ax.tick_params(axis = "x", labelsize = 16) ax.tick_params(axis = "y", labelsize = 16) # 軸のラベルをつける ax.set_xlabel("PC_1", fontsize = 20) ax.set_ylabel("PC_2", fontsize = 20) # データをプロットする ax.scatter(X_train_pca[y_train_class == 1, 0], X_train_pca[y_train_class == 1, 1], c = "dodgerblue", label = "AD_inside") ax.scatter(X_train_pca[y_train_class == -1, 0], X_train_pca[y_train_class == -1, 1], c = "crimson", label = "AD_outside") # Legend をつける ax.legend(loc = "upper left", fontsize = 18) # 図を保存 fig.savefig("../result/AD_plot.png")
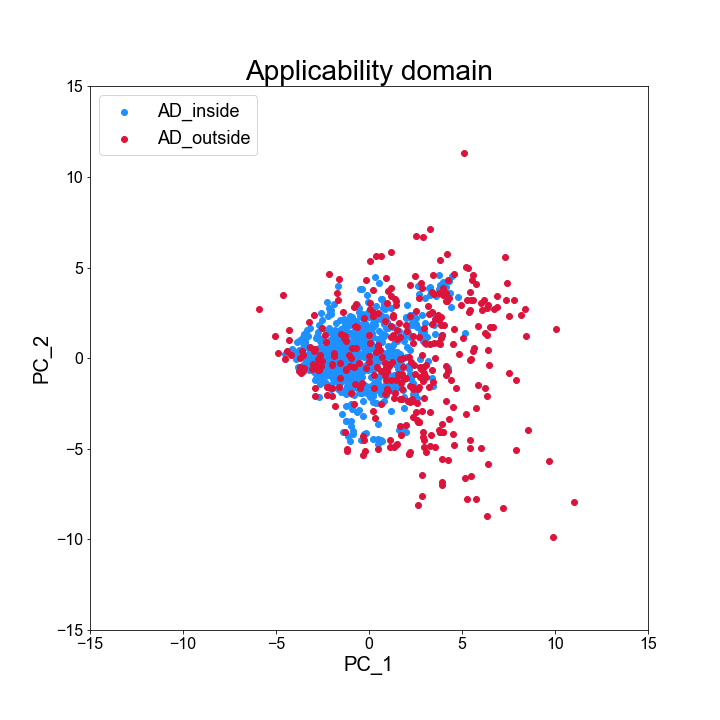
青(AD 内)は比較的原点に固まっているのに対し、赤(AD 外)は比較的原点から離れた領域に分散しているのが分かります。
続いて、テストデータについて、AD 内外で予測結果に影響するのか、y-y plot と R2 値を元に確認してみます。
# 線形重回帰モデル rgr = LinearRegression() rgr.fit(X_train, y_train) y_pred = rgr.predict(X_test) # y-y plot を作成 # フォントを変更 plt.rcParams["font.family"] = "Arial" # Plot のインスタンスと図の大きさを定義 fig = plt.figure(figsize = (10, 10)) # インスタンスの中に複数のグラフを作る場合に指定 # 引数は(行数、列数、具体的に作りたい図のインデックス) # グラフを 1 つだけ作りたいときは (1, 1, 1)としてやれば良い ax = fig.add_subplot(1, 1, 1) # グラフのタイトル ax.set_title("y-y plot", fontsize = 28) # グラフの表示範囲 ax.set_xlim(-9, 2) ax.set_ylim(-9, 2) # 目盛の数字の大きさを調整 ax.tick_params(axis = "x", labelsize = 16) ax.tick_params(axis = "y", labelsize = 16) # 軸のラベルをつける ax.set_xlabel("True_LogS", fontsize = 20) ax.set_ylabel("Pred_LogS", fontsize = 20) # 対角線(y = x)を引く ax.plot(list(range(-9, 3)), list(range(-9, 3)), "k") # データをプロットする ax.scatter(y_test, y_pred, c = "dodgerblue") # データをプロットする ax.scatter(y_test[y_test_class == 1], y_pred[y_test_class == 1], c = "dodgerblue", label = "AD_inside") ax.scatter(y_test[y_test_class == -1], y_pred[y_test_class == -1], c = "crimson", label = "AD_outside") # 図を保存 fig.savefig("../result/y_y_plot_withAD.png") # AD 内のデータについて R2 値を算出 print(r2_score(y_test[y_test_class == 1], y_pred[y_test_class == 1])) # AD 外のデータについて R2 値を算出 print(r2_score(y_test[y_test_class == -1], y_pred[y_test_class == -1]))
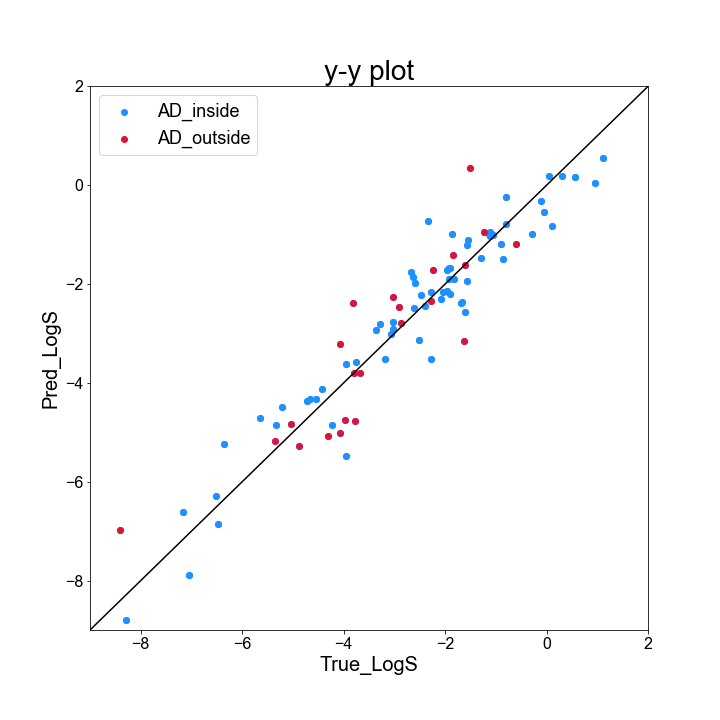
プロットを見ると、気持ち赤のプロットは対角線から離れているように見えますが、正直よく分かりません・・・
R2 値を見てみると、AD 内のデータが 0.920、AD 外のデータが 0.760 と、そこそこ差のある結果となり、AD 外のデータの予測が難しいことが見て取れます。
最後に、AD 内外の化学構造を確認してみます(コードはデータ構造がきれいではないので、解説はしないです)。
# テストデータに含まれる化学構造を表示 from rdkit.Chem.Draw import MolsToGridImage train_data_indices = pd.read_csv("../data/train_origin_tag.csv", index_col = 0) test_data_indices = pd.read_csv("../data/test_origin_tag.csv", index_col = 0) structures = Chem.SDMolSupplier("../data/logSdataset1290_2d.sdf") # AD 内に含まれるデータの構造を選択 test_structure_indices = [i.item() for i in np.array(test_data_indices)[y_test_class == -1][:20]] test_structure_examples = [structures[i] for i in test_structure_indices] # PNG 画像を作成し保存 img = MolsToGridImage(test_structure_examples, molsPerRow = 5, subImgSize = (300,200)) img.save("../result/test_outsideAD.png")
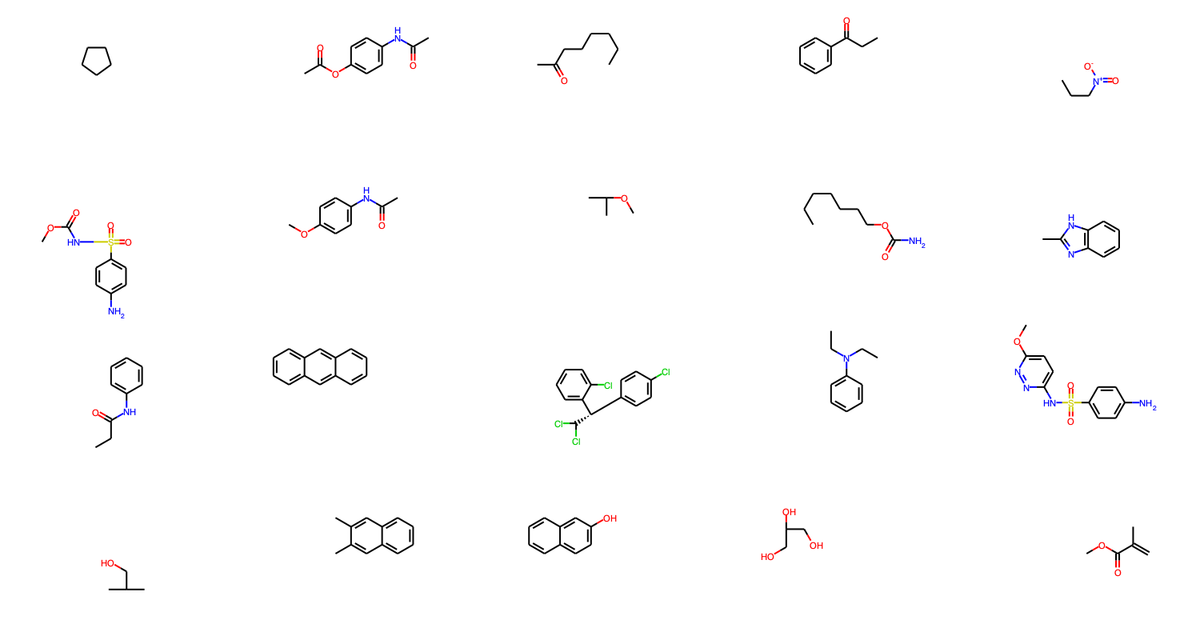
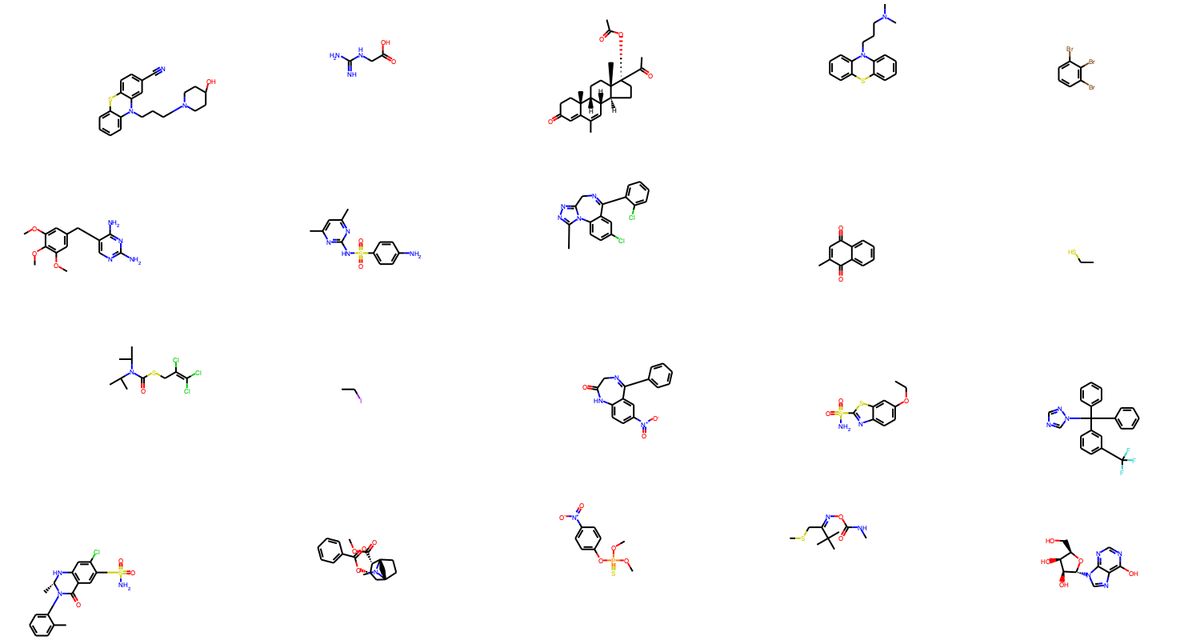
AD 外に含まれる構造は、AD 内に含まれる構造よりも複雑で、外れ値となりそうなのが化学的にも見て取れると思います。
AD を考慮することで、より適切な機械学習モデルの運用が実現すると思います。